您所在的位置:
作物有害生物功能基因组研究创新团队建立新型水稻基因组编辑工具盒-CRISPR/ Sc++系统
来源:郑州天顺食品添加剂有限公司
发布时间:2021-09-01 09:35:47
关注: 0 次
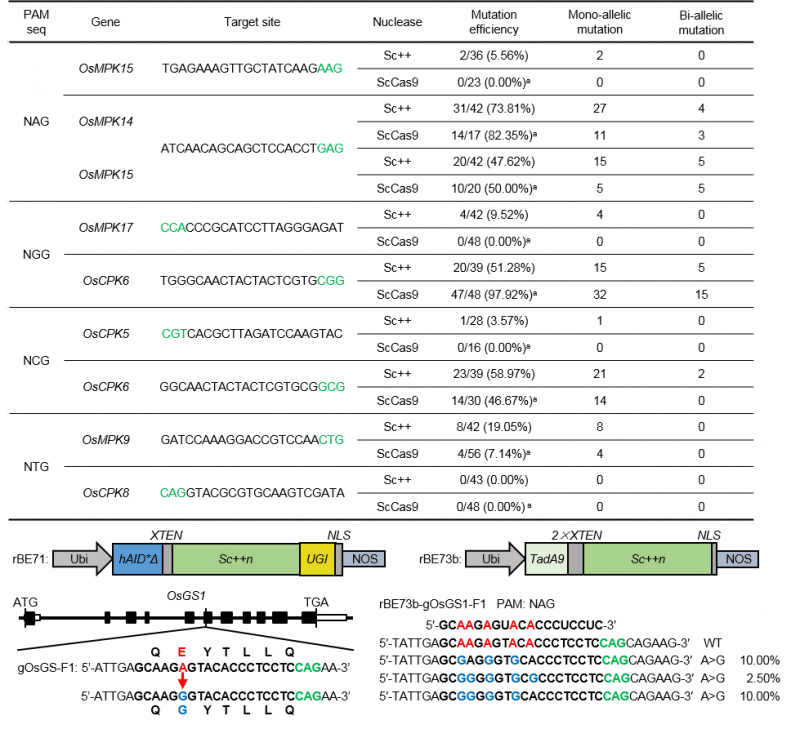
近日,中国农业科学院植物保护研究所作物有害生物功能基因组研究创新团队在国际知名学术期刊《植物学报(Journal of Integrative Plant Biology)》在线发表了题为“CRISPR/Sc++ system-mediated genome modification in rice” 的研究论文。该论文报道了新型的CRISPR/ Sc++系统在水稻基因组编辑中的应用,Sc++突变体可突破野生型ScCas9的位点依赖性,高效识别NNG PAN完成靶基因的编辑,扩宽了水稻基因组编辑的应用范围。
CRISPR/Cas9系统已成为基因组精准修饰的有效工具,有效促进了水稻功能基因组学研究和分子育种进程。然而Cas蛋白需识别特定的PAM序列, 对基因编辑靶点具有选择性,因此扩展Cas9的识别范围是当前对CRISPR/Cas9系统(尤其是碱基编辑系统)优化改良的重要方向。利用不同物种Cas9蛋白及其突变体等可在一定程度上扩宽CRISPR工具的打靶范围。例如,应用最广泛的化脓性链球菌Cas9 (SpCas9)识别DNA靶点下游保守的NGG PAM序列。而SpCas9的突变体,如SpCas9-NG、xCas9、SpG、SpRY等(Ren et al., 2019; Xu et al., 2021),这些蛋白经改造后也具有不同或更为灵活的PAM兼容性,在一定程度拓宽了基因组编辑工具的靶向范围。因此,开发识别PAM灵活和编辑效率高的Cas蛋白,对于拓展CRISPR/Cas9系统的应用范围具有重要的意义。
该研究通过对ScCas9蛋白进行优化,利用获得的突变体Sc++对水稻碱基编辑工具进行了优化升级。与ScCas9相比, Sc++核酸酶具有更广泛和高效的编辑能力,在NNG(NAG、NGG、NCG和NTG)PAM处均具有很好的编辑效率。基于Sc++和高效的胞嘧啶脱氨酶hAID*Δ和腺嘌呤脱氨酶TadA9,该团队开发了水稻胞嘧啶碱基编辑器rBE71和腺嘌呤碱基编辑器rBE73b。对转基因水稻的检测显示,rBE71和rBE73b均能通过识别NGG、NAG、NCG等PAM完成碱基编辑,效率分别高达39.13%和95.74%,还产生了除草剂抗性基因OsGS1的新等位基因。CRISPR/ Sc++系统有效地扩展了水稻基因组编辑工具应用范围,为后续基因组编辑衍生工具开发、水稻功能基因组学研究和分子设计育种提供了有力的理论指导和技术支撑。
中国农业科学院植物保护研究所科研助理马桂根硕士和博士生旷永洁为论文共同第一作者,周焕斌研究员为通讯作者,团队多名成员共同合作完成。该研究得到了中国国家自然科学基金和中央级公益性科研院所基本科研业务费专项的项目支持。
论文链接:https://doi.org/10.1111/jipb.13166
文章版权备注
文章版权归 郑州天顺食品添加剂有限公司 所有
文章链接:https://www.tsswhg.com/4867.html
未经授权,禁止任何站点镜像、采集、或复制本站内容,违者通过法律途径维权到底!
【推荐阅读】
- 2023-05-01奶味香精的制备技术与开发现状
- 2023-05-01菌种强化结合工艺优化提高酱香白酒基酒中四甲基吡嗪含量的研究(二)
- 2023-05-01发酵小麦胚芽产2,6-二甲氧基对苯醌菌种筛选及发酵条件优化(一)
- 2023-04-30茵栀黄化学成分及药理作用研究进展(一)
- 2023-04-04阿魏酸及其衍生物在食品添加剂领域研究进展(一)
- 2023-03-23“和美魏县,梨享天下”魏县鸭梨产业发展研讨会圆满落幕
- 2023-03-23关于参加山东食博会开幕式及餐饮高峰论坛的通知
- 2023-03-23超90%展商已确认展位 6月食品原料展带来海内外商机
- 2023-03-23关于召开2023年国际食品安全与健康大会的通知
- 2023-03-23白俄罗斯下调对中国等国家乳制品最低出口限价

 豫ICP备19024296号
豫ICP备19024296号 售前咨询
售前咨询